1. 安装
在主菜单找到Apps manager,再找到BinGO就可以实现安装
2. 使用
使用之前先下载物种最新的GO注释文件,以拟南芥为例:
http://geneontology.org/page/downloads
找到拟南芥,这个注释文件包含的两列对这里的分析是有用的,分别是Gene ID和GO 功能注释。但文件还包含了其他信息,如symbol,基因的物理坐标信息,UniProt ID等:
将刚刚下载的gene_association.tair导入到BiNGO中.
下载GO功能注释分类文件,下载地址:http://geneontology.org/page/downloads
相同界面,点选Ontology 在界面内,找到“go-basic.obo”并下载
“go-basic.obo”可以使用文本文件打开。里面记录了GO term 间的关系,依据这些关系,GO term最终将被化成网络图的形式。找到 select ontology file的窗口,选择custom,然后导入。
针对拟南芥,也可以使用cytoscape自带的注释和注释分类文件,不需要重新下载
3. 结果
GO富集结果:一个文件属于弹窗,另一个存储在输出文件夹中(内容相同)。第五列和第六列列出了这类功能基因在目标基因集合和全基因组基因中的比例。
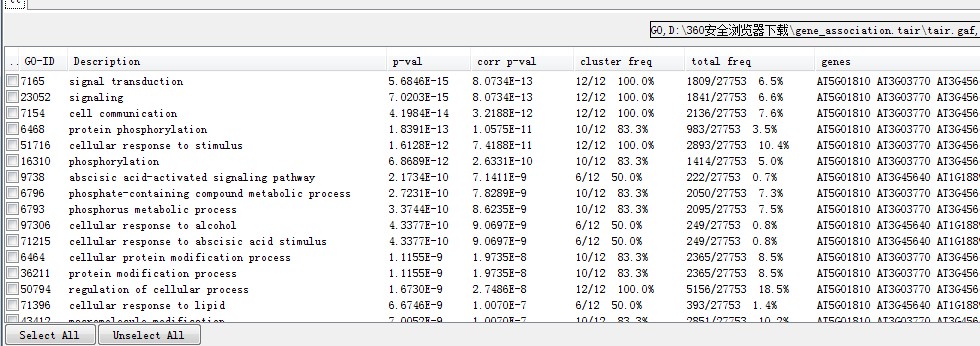
另外GO分析网络图结果,颜色越深越富集,下面的表格为详细的富集信息:
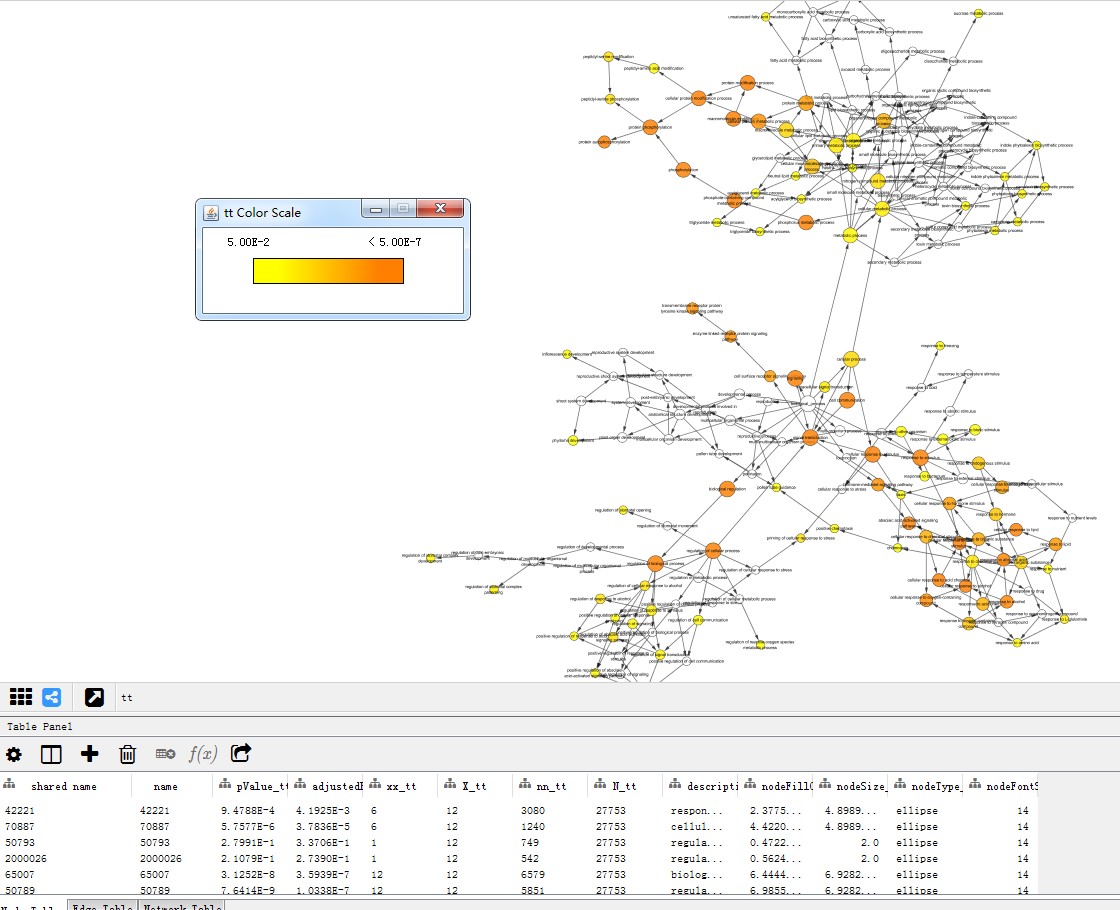
上述步骤分析完后会直接在cytoscape 页面生成绘图结果
最后一次修改于 2018-12-17